Überblick
INSIDe wird eine neue Plattform für die integrative datengesteuerte Analyse der Ausbreitung von Infektionskrankheiten entwickeln. Dazu bringt das Konsortium führende Experten aus den Bereichen mathematische und rechnergestützte Modellierung, Epidemiologie und Erforschung von Infektionskrankheiten zusammen. INSIDe wird die von den Partnern unabhängig voneinander entwickelten Ansätze kombinieren und weiterentwickeln, um eine flexible Integration verschiedener Datenquellen zu ermöglichen, darunter (i) amtliche Fall-, Sterbe-, Impf- und Hospitalisierungsstatistiken, (ii) repräsentative Kohortenstudien und (iii) Abwasserproben. Der Einsatz von Simulationsmodellen mit mehreren Auflösungen und umfassenden Modellen für Beobachtungsprozesse wird eine eingehende Bewertung und die Untersuchung großer Regionen ermöglichen.
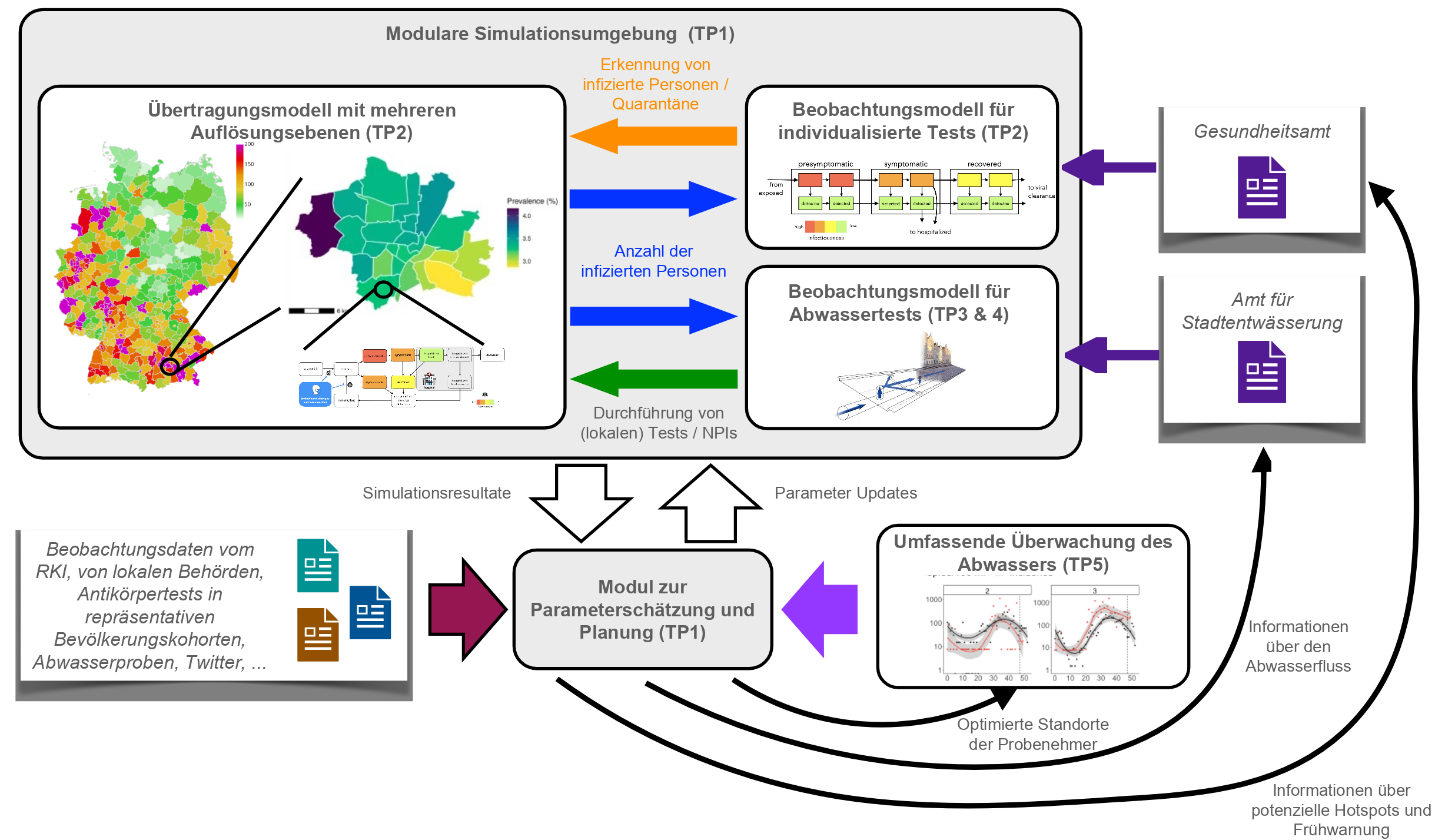
Die technologische Grundlage von INSIDe sind die drei komplementären, hochmodernen Software-Frameworks pyABC [1], MEmilio [2, 3] und ++SYSTEMS [4], die von der Universität Bonn, dem Deutschen Zentrum für Luft- und Raumfahrt bzw. Tandler entwickelt wurden.
- pyABC ermöglicht die datengetriebene Modellierung von Mehrskalenprozessen. In AP1 wird es um ML-Methoden erweitert, um die sequentielle Aktualisierung von Parameterschätzungen in Längsschnittstudien zu ermöglichen.
- MEmilio ermöglicht die Simulation der räumlich-zeitlichen Ausbreitung von Infektionskrankheiten. In AP2 wird es um eine Multiresolution-Simulationsfunktionalität erweitert, um die Untersuchung von Prozessen zu erleichtern, die auf verschiedenen Skalen stattfinden, und mit Modellen für den Beobachtungsprozess ausgestattet.
- ++SYSTEMS ermöglicht die feinmaschige Simulation von Strömungsmustern in Abwassersystemen durch die Universität der Bundeswehr München (AP3). In AP4 wird es erweitert, um die effiziente Erstellung grobkörniger Modelle für größere Einzugsgebiete zu ermöglichen.
Für die Parametrisierung und Validierung der verbesserten Modelle und der integrierten Pipeline werden wir veröffentlichte und unveröffentlichte Daten nutzen. Am wichtigsten ist der einzigartige Zugang zu den Daten der repräsentativen Bevölkerungs- und Abwasserstichprobenstudien in (i) München, Deutschland, und (ii) Addis Abeba und Jimma, Äthiopien, die in von der KUM koordinierten Projekten erhoben werden. Die verfügbaren Daten werden durch zusätzliche Stichprobenerhebungen ergänzt (AP5).
Die INSIDe-Plattform wird die Bewertung des Zustands von Epidemien und Pandemien, die Analyse ihrer Dynamik und die Vorhersage des weiteren Verlaufs erleichtern. Darüber hinaus wird sie eine datengestützte Bewertung von nicht-pharmazeutischen Maßnahmen ermöglichen, die von einem krankheits- und Erregerstammübergreifenden Ansatz profitieren. Dazu werden mechanistische Beschreibungen der Interventionen in das Modell implementiert und die Effektgrößen anhand des kombinierten Informationsgehalts der verschiedenen vorgenannten Datenquellen geschätzt. Die explizite Beschreibung des Beobachtungsprozesses (und damit der zeitabhängigen Wahrscheinlichkeit, ein infiziertes Individuum zu entdecken) wird die Entschlüsselung von Effekten erleichtern, die von NPM (z.B. Maskentragen, Quarantäne, Schließung von Veranstaltungsorten usw.) und Änderungen der Beobachtungsprozesse, z.B. Testverfügbarkeit und -strategie, ausgehen.
[1] Klinger E., Rickert D., Hasenauer J. pyABC: distributed, likelihood-free inference. bioRxiv. 2017; 162552.
[2] Kühn M.J., Abele D., Mitra T., Koslow W., Abedi M., Rack K., et al. MEmilio - a high performance Modular EpideMIcs simuLatIOn software. Available: https://github.com/DLR-SC/memilio.
[3] Kühn M.J., Abele D., Mitra T., Koslow W., Abedi M., Rack K., et al. Assessment of effective mitigation and prediction of the spread of SARS-CoV-2 in Germany using demographic information and spatial resolution. Math Biosci. 2021;339: 108648.
[4] tandler.com GmbH. ++Systems Isar. tandler.com GmbH; 18 Feb 2020 [cited 4 Oct 2021]. Available: https://tandler.com/de/page/software/plus-plus-systems.
